Rの練習 #
0. 関連チュートリアル #
1. Rを使ってデータの読み込みをしてみよう #
解析環境を使ってみようで作ったプロジェクトの中でRを使ってみましょう。
- プロジェクトの作成方法
prem playground cursor playgroundこのコマンドで
playgroundという名前のプロジェクトを作成し、Cursorでそのプロジェクトディレクトリを開きます。
1.1. R scriptを作成する #
まずR言語を使用するためにR script(拡張子が.Rのファイル)を作成します。
touch code/practice.R
codeというディレクトリの中にpractice.RというR言語のプログラムを記載するためのファイルが作成されます。
- 左のExplorer (エクスプローラー) ビューで、Projectディレクトリ(例: lecture1)を右クリックし、「New File…」を選択してファイル名を指定する。
CursorのGUIを使ってより直感的な操作も可能です。
1.2. パッケージのインストール #
Rのパッケージは、特定の機能(データ読み込み、統計解析、グラフ作成など)を提供してくれる拡張機能のようなものです。renv を使っているプロジェクトでは、renv::install() 関数を使ってパッケージをインストールするのが一般的です。
ここでは例として、CSVファイルを簡単に読み込むためのreadrパッケージをインストールしてみましょう。
practice.R ファイルに以下のコードを追加してください。
1# readr パッケージをインストール
2renv::install("readr")コードを追加したら、追加した部分にカーソルを合わせて Ctrl+Enter (Windows), Cmd+Enter (Mac) を押して実行します。
インストールが完了すると、コンソールにメッセージが表示されます。renv はプロジェクトごとにインストールしたパッケージを管理してくれるため、他のプロジェクトに影響を与えません。
1.3. インストールしたパッケージを使ってみる #
readr パッケージを使って、簡単なデータ読み込みのデモを行います。まず、試しに読み込むためのCSVファイルを作成しましょう。
Projectディレクトリ(playground)内の、dataというディレクトリの中にsample_data.csv という名前で新しいファイルを作成し、以下の内容をコピーアンドペーストしてください。
1gene_name, sample1, sample2, sample3
2 gene1, 10, 20, 40
3 gene2, 15, 40, 35
4 gene3, 30, 25, 45
5 gene4, 25, 35, 50
6 gene5, 20, 30, 30次に、practice.R ファイルに以下のコードを追加します。
# readr パッケージを読み込みます
library(readr)
# sample_data.csv ファイルを読み込みます
data <- read_csv("data/sample_data.csv")こちらもコードを追加したら、追加した部分にカーソルを合わせて実行してみてください。
library(readr)は、インストールしたreadrパッケージを現在のRセッションで使用可能にするためのコードです。- パッケージを使う前には必ず
library()で使いたいパッケージを読み込む必要があります。
次に読み込んだデータを表示してみます。
print(data)# A tibble: 5 × 4
gene_name sample1 sample2 sample3
<chr> <dbl> <dbl> <dbl>
1 gene1 10 20 40
2 gene2 15 40 35
3 gene3 30 25 45
4 gene4 25 35 50
5 gene5 20 30 30これは readr パッケージの read_csv 関数を使ってファイルが正しく読み込めたことを示しています。
- このような
dataをtibbleと言います。- 列名がついた列 と 行名がついていない行 からなるデータ形式です。
1.4. コンソールでデータを確認する。 #
データの中から一部の行や列、値を取り出してみましょう。
# 最初の3行を表示
head(data, 3)# A tibble: 3 × 4
gene_name sample1 sample2 sample3
<chr> <dbl> <dbl> <dbl>
1 gene1 10 20 40
2 gene2 15 40 35
3 gene3 30 25 45# gene_name列を取り出す
print(data$gene_name)[1] "gene1" "gene2" "gene3" "gene4" "gene5"# sample1列を取り出す
print(data$sample1)[1] 10 15 30 25 20# 列番号で列を取り出す
print(data[, 1])# A tibble: 5 × 1
gene_name
<chr>
1 gene1
2 gene2
3 gene3
4 gene4
5 gene5 # 行番号で行を取り出す
print(data[1, ])# A tibble: 1 × 4
gene_name sample1 sample2 sample3
<chr> <dbl> <dbl> <dbl>
1 gene1 10 20 40# 特定の値を取り出す
print(data[1, 2])# A tibble: 1 × 1
sample1
<dbl>
1 10# 特定の範囲の行と列を取り出す
print(data[1:2, 2:4])# A tibble: 2 × 3
sample1 sample2 sample3
<dbl> <dbl> <dbl>
1 10 20 40
2 15 40 352. グラフを作成してみよう #
Rではグラフを作成するための関数がたくさん用意されています。
特にggplot2というパッケージは非常にきれいなグラフを作成することができます。
多くの論文でこのパッケージが使用されているため、これを使えば論文で見たことのあるFigureを作成することができます!
tutorialsのggplot2も参考にしながら練習してみましょう。
2.1. ggplot2のインストール #
ggplot2パッケージをインストールします。
renv::install("ggplot2")2.2. bar plotを作成してみよう #
# plot用のlibraryを読み込む
library(ggplot2)
# sample1 の bar plot を作成
ggplot(data, aes(x = gene_name, y = sample1)) +
geom_bar(stat = "identity")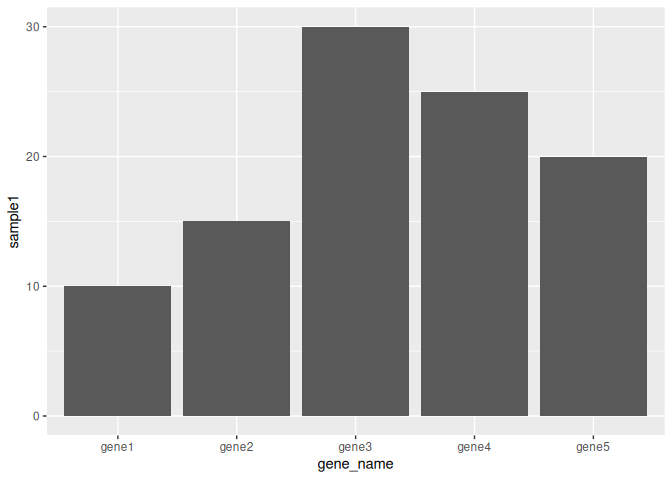
# sample2 はすこし見た目を変える
ggplot(data, aes(x = gene_name, y = sample2)) +
geom_bar(stat = "identity") +
theme_minimal()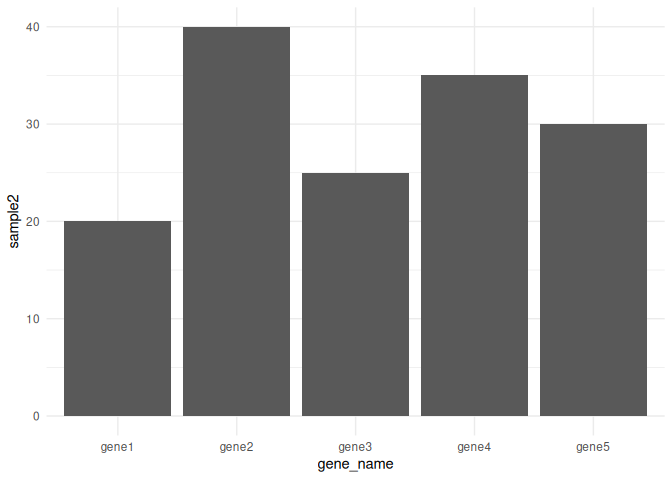
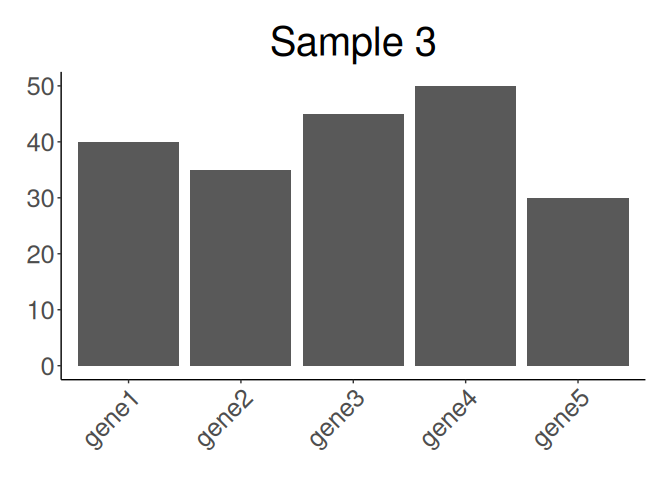
# sample3 はもっと細かく見た目をいじる
ggplot(data, aes(x = gene_name, y = sample3)) +
geom_bar(stat = "identity") +
ggtitle("Sample 3") +
theme_classic() +
theme(
text = element_text(size = 24),
plot.title = element_text(size = 30, hjust = 0.5),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1),
plot.margin = margin(t = 20, b = 20, r = 20, l = 20, unit = "pt")
)
- 他にもバーの色を変えたり、バーに枠線をつけたり、y軸の0をx軸と一致させたり・・・
- AI chatでコードを渡しながら「バーの色を変えたい」「y軸の0とx軸を一致させたい」と聞くと・・・
2.3. グラフの保存 #
グラフを保存するには、ggplot オブジェクトを ggsave 関数に渡します。
- 一旦グラフを変数に保存してから保存します。
- 変数に入れなくても、最後に実行したggplotオブジェクトは直後にggsaveで保存できます。
bar_plot <- ggplot(data, aes(x = gene_name, y = sample3)) +
geom_bar(stat = "identity") +
ggtitle("Sample 3") +
theme_classic() +
theme(
text = element_text(size = 24),
plot.title = element_text(size = 30, hjust = 0.5),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1),
plot.margin = margin(t = 20, b = 20, r = 20, l = 20, unit = "pt")
)
# グラフを保存
ggsave("bar_plot.png", bar_plot, width = 10, height = 8)
ggsave関数は、グラフをPNG、PDF、JPEGなどの形式で保存することができます。
3. AIに聞きながらグラフを作成してみる #
- ここから先は一部のコードを隠しておきます。
- 画像だけ見てAIに聞いてコードを作れるか練習してみましょう。
- 一度質問をしてからコードを見ると「AIにこう聞けば良かったのかも」のような発見があるかもしれません。
3.0. AI chatの使い方 #
ctrl+L(win) orcmd+L(mac)でチャットエリアを開くことができます。- チャットエリアを開いたときにエディットエリアで編集中のファイル(今回だと
practice.R)が自動的にコンテクストとして認識されます。 - つまり自動的に今書いているコードを読んで、ユーザーが次に何をしたいかを推測しながらコードを生成してくれます。
- コードのなかの「特にこの部分が知りたい」という時には、その部分をドラッグで選択してから
ctrl+L(win) orcmd+L(mac)を押すと、その部分をコンテクストとして認識してくれます。 - 詳しくはCursor - AI Chatでコードを書くを参考にしてください。
3.1. scatter plot を作成してみる #
# scatter plot を作成
ggplot(data, aes(x = sample1, y = sample2)) +
geom_point()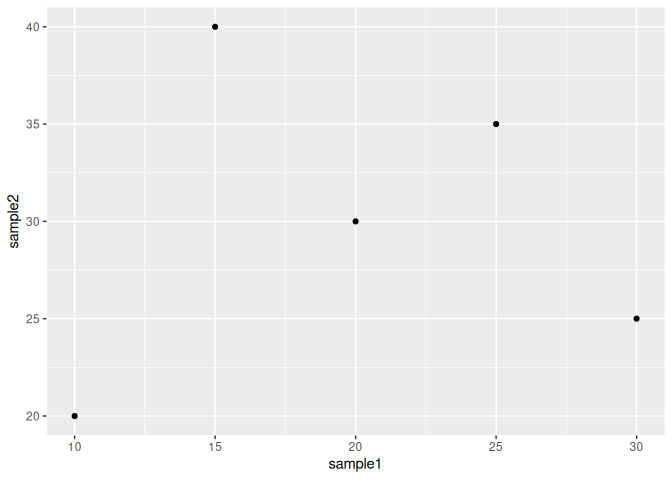
ラベルを加えて、点と文字を大きくし、背景を白にする
ggplot(data, aes(x = sample1, y = sample2)) +
geom_point(size = 6, alpha = 0.5) +
geom_text(aes(label = gene_name), size = 8, vjust = -1, hjust = -0.1) +
theme_classic() +
theme(
text = element_text(size = 24),
axis.title.x = element_text(size = 24, vjust = -3),
axis.title.y = element_text(size = 24, vjust = 5),
plot.margin = margin(t = 60, r = 60, b = 60, l = 60, unit = "pt")
) +
coord_cartesian(clip = "off")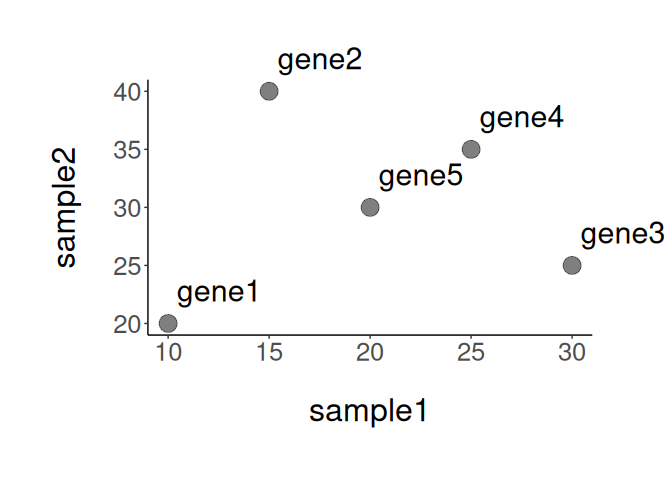
3.2. heatmap を作成してみる #
# heatmap用のきれいなカラーパレットを読み込む
library(RColorBrewer)
# 色一覧を表示
display.brewer.all()
heatmapにするには行名と列名がついた行列データが必要なので、データを行列に変換
# heatmapにするには行名と列名がついた行列データが必要
matrix_data <- as.matrix(data[, -1])
rownames(matrix_data) <- data$gene_name
# データを表示
print(matrix_data) sample1 sample2 sample3
gene1 10 20 40
gene2 15 40 35
gene3 30 25 45
gene4 25 35 50
gene5 20 30 30# heatmap を作成
heatmap(matrix_data,
col = brewer.pal(11, "RdBu"),
scale = "row",
margins = c(10, 10))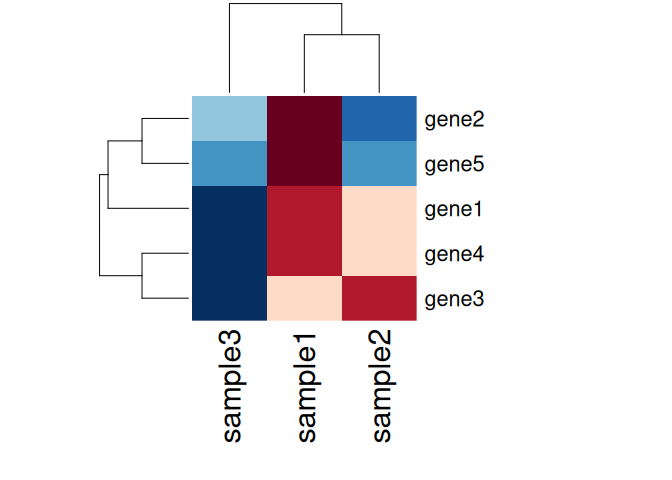
4. tidy dataを使ったグラフ作成 #
tidy data(整然としたデータ)は、データ解析において非常に重要な概念です。
チュートリアルに詳しい説明がありますが、ひとまず機械にとって解析しやすいデータ形式と覚えておけば良いです。
tidyverseのチュートリアルも参考にしながら練習してみましょう。
4.1. tidy data(縦長データ)に変換する #
- 一つの変数は一つの列に(Each variable must have its own column.)
- 一つの観測は一つの行に(Each observation must have its own row.)
- 一つの値は一つのセルに(Each value must have its own cell.)
このルールに従って変換したデータがtidy dataです。
次の例を見るとわかりますが、この原則を守るとデータは縦長になります。
# tidy data(縦長データ)に変換するためのlibraryを読み込む
library(tidyr)# tidy data(縦長データ)に変換
data_tidy <- data |>
pivot_longer(cols = -gene_name, names_to = "sample", values_to = "value")
# 変換後のデータを表示
print(data_tidy)# A tibble: 15 × 3
gene_name sample value
<chr> <chr> <dbl>
1 gene1 sample1 10
2 gene1 sample2 20
3 gene1 sample3 40
4 gene2 sample1 15
5 gene2 sample2 40
6 gene2 sample3 35
7 gene3 sample1 30
8 gene3 sample2 25
9 gene3 sample3 45
10 gene4 sample1 25
11 gene4 sample2 35
12 gene4 sample3 50
13 gene5 sample1 20
14 gene5 sample2 30
15 gene5 sample3 304.2. サマリを作ってみる #
tidy dataは非常に計算しやすいです。
簡単に平均値や標準偏差といった統計量を計算できます。
# summary を計算するためのlibraryを読み込む
library(dplyr)遺伝子の平均値と標準偏差を計算
# 遺伝子の平均値と標準偏差を計算
data_summary <- data_tidy |>
group_by(gene_name) |>
summarise(mean = mean(value), sd = sd(value))
# 結果を表示
print(data_summary)# A tibble: 5 × 3
gene_name mean sd
<chr> <dbl> <dbl>
1 gene1 23.3 15.3
2 gene2 30 13.2
3 gene3 33.3 10.4
4 gene4 36.7 12.6
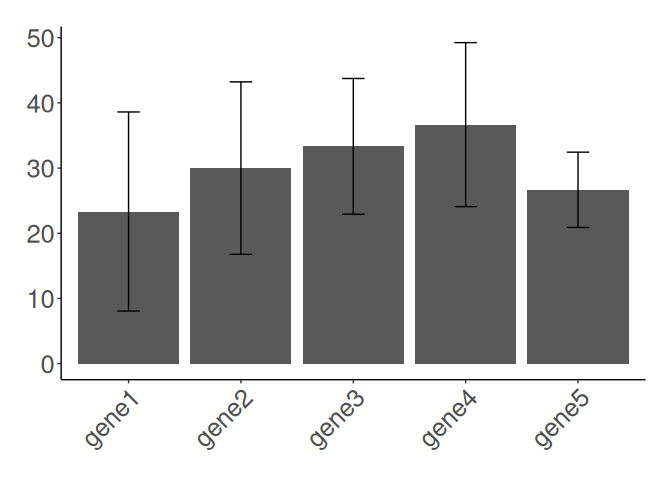
5 gene5 26.7 5.774.3. summaryを使ってbar plot を作成してみる #
先ほど作成したbar plotとは違って、各サンプルの情報が集計されたsummaryがあります。
これを使えば複数サンプルからなるデータの平均とばらつきを視覚で表現できます。
平均値と標準偏差をそれぞれbarとerror barで表示してみましょう。
summaryを使ってbar plotを作成
# summaryを使ってbar plotを作成
ggplot(data_summary, aes(x = gene_name, y = mean)) +
geom_bar(stat = "identity") +
geom_errorbar(aes(ymin = mean - sd, ymax = mean + sd), width = 0.2) +
theme_classic() +
theme(
text = element_text(size = 24),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1),
plot.margin = margin(t = 20, b = 20, r = 20, l = 20, unit = "pt")
)
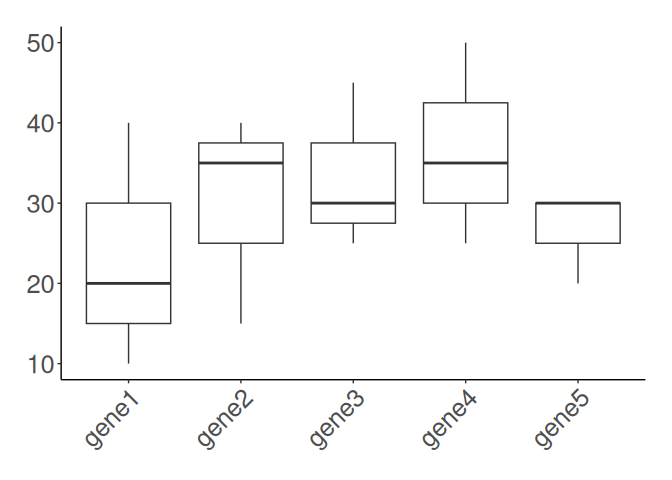
4.4. box plot を作成してみる #
データのばらつきを表現するもう一つの方法としてbox plotがあります。
中央値、四分位数、最小最大値を表示することでデータのばらつきをより直感的に表現できます。
# box plot を作成
ggplot(data_tidy, aes(x = gene_name, y = value)) +
geom_boxplot() +
theme_classic() +
theme(
text = element_text(size = 24),
plot.title = element_text(size = 30, hjust = 0.5),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1),
plot.margin = margin(t = 20, b = 20, r = 20, l = 20, unit = "pt")
)
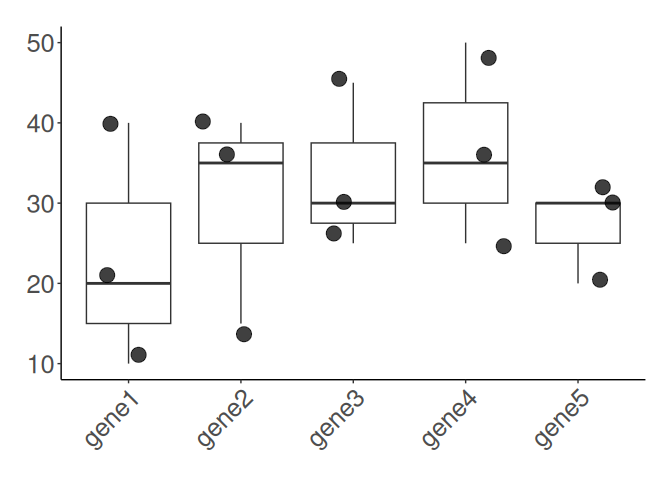
4.5. box dot plot を作成してみる #
さらにdot plotやjitter plotによってデータ点を加えることによって、データのばらつきをさらに直感的に表現できます。
box dot plot を作成
# box dot plot を作成
ggplot(data_tidy, aes(x = gene_name, y = value)) +
geom_boxplot(outlier.shape = NA) +
geom_jitter(size = 5, alpha = 0.75) +
theme_classic() +
theme(
text = element_text(size = 24),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1),
plot.margin = margin(t = 20, b = 20, r = 20, l = 20, unit = "pt")
)